27.06.2008
Werkbank für Antibiotika
Marburger Wissenschaftler erforschen, wie bioaktive Peptide synthetisiert werden
Biochemiker von der Philipps-Universität haben die Struktur einer molekularen Werkbank aufgeklärt, mit der Bakterien eine Vielzahl von Peptiden herstellen, darunter auch wichtige Antibiotika und Fungizide. Wie die Wissenschaftler um Professor Dr. Lars-Oliver Essen und Professor Dr. Mohamed A. Marahiel in der aktuellen Online-Ausgabe von „Science“ berichten, bilden die untersuchten Moleküle eine Plattform, auf der Aminosäuren zu Ketten zusammengebaut werden. „Die Kenntnis dieses Mechanismus’ eröffnet neue Möglichkeiten für die Produktion von Arzneimitteln“, erläutert Professor Essen.
Peptide sind kurze Ketten aus Aminosäuren, im Unterschied zu den langkettigen Proteinen. Mikroorganismen können Peptide in weitaus größerer struktureller Vielfalt produzieren als Proteine, für deren Herstellung komplex aufgebaute Zellbestandteile benötigt werden, so genannte Ribosomen. Peptide erfüllen mannigfaltige biologische Funktionen und kommen deshalb für die Entwicklung von neuen Wirkstoffen in Frage. Die Einzeller bewerkstelligen die Pepdidsynthese mittels spezialisierter Enzymkomplexe, so genannter Nichtribosomaler Peptidsynthetasen (NRPS). Durch ihren modularen Aufbau arbeiten sie wie ein Fließband, auf dem die Peptide aus Aminosäuren zusammengesetzt werden. Jedes Modul des Enzyms übernimmt dabei eine bestimmte Funktion.
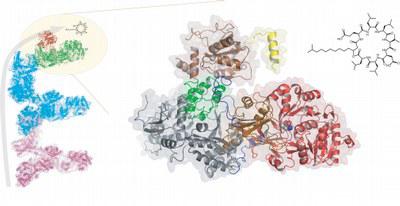
Die so genannte PCP-Domäne ist als dritte Untereinheit flexibel an die Plattform gebunden, so dass sie mitsamt der an sie angehefteten Aminosäure zwischen die beiden Module der Werkbank schwenken kann. Die vierte Untereinheit ist für die Freisetzung des vollständigen Peptidantibiotikums verantwortlich. „Die Struktur dieses modularen Enzyms macht deutlich, wie die aufeinander folgenden katalytischen Schritte koordiniert werden“, fassen die Wissenschaftler ihre Ergebnisse zusammen.
Der Vorteil dieser Art der Peptidsynthese liegt unter anderem darin, dass hierbei auch unkonventionelle Aminosäuren als Bausteine verwendet werden können, die in den körpereigenen Eiweißstoffen nicht vorkommen. Dadurch stehen weit mehr als die herkömmlichen 21 Aminosäuren zur Verfügung, so dass eine wesentlich größere Vielfalt an Verbindungen möglich ist. „Die ermittelte Kristallstruktur regt zu weiteren Untersuchungen an, um die molekularen Fließbänder gezielt umzugestalten, so dass sie zur Produktion neuartiger Wirkstoffe auf Peptidbasis dienen“, schreiben die Autoren des „Science“-Artikels.
Originalveröffentlichung:
Alan Tanovic, Stefan A. Samel, Lars-Oliver Essen & Mohamed A.
Marahiel:
Crystal Structure of the Termination Module of a Nonribosomal
Peptide Synthetase.
www.sciencexpress.org
DOI: 10.1126/science.1159850
Weitere Informationen:
Ansprechpartner:
Professor Dr. Lars-Oliver Essen
Fachgebiet Biochemie
Tel.: 06421
28-22032
Internet:
http://www.uni-marburg.de/fb15/ag-essen
Prof. Dr. Mohamed A. Marahiel
Tel.:
06421 28-25722