20.07.2012
Wie der Wirt Bakterien erkennt
Internationales Wissenschaftlerteam erforschte das angeborene Immunsystem
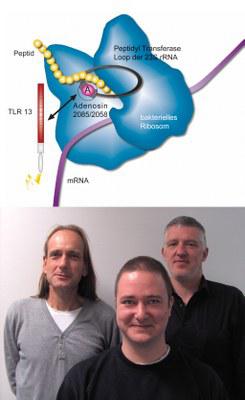
(Abbildung: Daniela Willemsen)
Die Marburger Immunologen (unten, von links) Dr. Andreas Kaufmann, Gernot Nees und Professor Dr. Stefan Bauer sind an der Veröffentlichung in „Science“ beteiligt.
(Foto: Philipps-Universität Marburg / AG Bauer)
Unser angeborenes Immunsystem ist klug. Es erkennt bestimmte Strukturen, die charakteristisch für viele Mikroorganismen sind, und schlägt Alarm. Toll-like Rezeptoren lösen dann eine Entzündungsreaktion aus. Das kann aber auch gefährlich werden: Weil TLR so empfindlich sind, kommt es bei schweren Infektionen zum Überschießen der Immunantwort. Die Folge: Blutvergiftung und oftmals ein septischer Schock.
Deshalb nahmen die Experten um Professor Dr. Carsten Kirschning von der Universität Duisburg-Essen und Professor Dr. Stefan Bauer von der Philipps-Universität die Wirtserkennung von Staphylokokken - und E.coli -Bakterien, die häufig Blutvergiftungen auslösen, unter die Lupe: Sie konnten nachweisen, dass TLR13 ein spezifisches Segment der bakteriellen ribosomalen Ribonukleinsäure erkennt. Daran binden sich auch spezifische Antibiotika wie zum Beispiel Erythromycin, falls das Segment nicht wie bei resistenten Erregern verändert ist. Die konkrete Ribonukleinsäure wird als 23S rRNA bezeichnet. „Tierische und menschliche Ribosomen binden Erythromycin hingegen nicht an ihre 28S rRNA, weil ihre Struktur diesbezüglich der von 23S rRNA resistenter Bakterien ähnelt“, erklärt Kirschning.
Bedeutsam sind die neuen Erkenntnisse für die Behandlung bakterieller Infektionen und das Verständnis von Antibiotika-Resistenzen. Außerdem könnten sie bei der Therapie von immunologischen Überreaktionen helfen und neue Impfstrategien eröffnen.
Die Studie stammt von einem internationalen
Forscherteam um Professor Dr. Carsten Kirschning vom Institut für
Medizinische Mikrobiologie des Universitätsklinikums Essen der UDE,
Privatdozent Dr. Hubertus Hochrein von der Firma Bavarian Nordic GmbH
in Martinsried, Professor Dr. Stefan Bauer vom Institut für Immunologie
der Phillips-Universität und Professor Dr. Hermann Wagner vom Institut
für Medizinische Mikrobiologie, Immunologie und Hygiene der Technischen
Universität München. Dazu gehören weitere Wissenschaftler aus München,
der Schweiz und Japan. Die Marburger Arbeitsgruppe behandelte unter
anderem 23S ribosomale RNA aus Bakterien mit speziellen Enzymen, die
einen bestimmten Baustein der RNA chemisch modifizieren, so dass diese
nicht von TLR13 erkannt wird.
(Pressetext: Carmen Tomlik, Universität Duisburg-Essen)
Originalveröffentlichung: Marina Oldenburg, Anne Krüger, Ruth Ferstl & al.: TLR13 recognizes bacterial 23S rRNA devoid of erythromycin resistance-forming modification, Science 19. Juli 2012
Weitere Informationen:
Ansprechpartner: Professor Dr. Stefan Bauer,
Institut für Immunologie
Tel.: 06421 28-65315
E-Mail:
bauerst@staff.uni-marburg.de
Professor Dr. Carsten Kirschning,
Universitätsklinikum Essen
Tel.: 0201 723-1824
E-Mail:
carsten.kirschning@uk-essen.de