03.11.2017 Kristallografie klärt Herstellung von Eisen-Schwefel-Clustern
Internationales Team deckt 3D-Struktur eines Proteinkomplexes der Mitochondrien auf
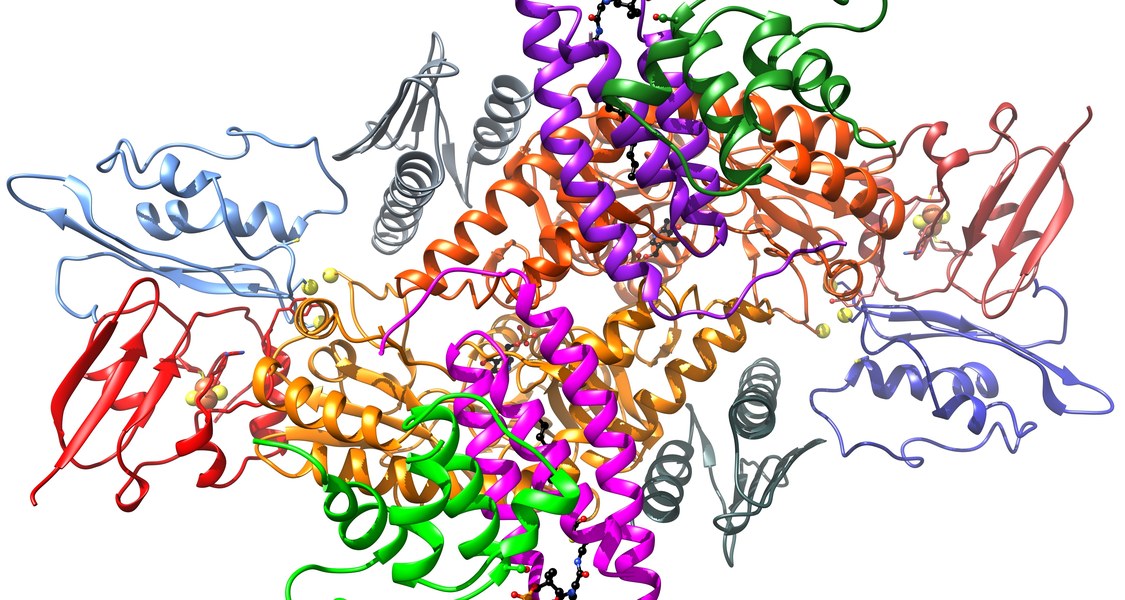
Wissenschaftler aus Marburg und Kanada haben die Struktur eines Proteinkomplexes aufgeklärt, der für die Herstellung lebenswichtiger Enzyme erforderlich ist. Die Forschungsgruppe um den Marburger Biochemiker Professor Dr. Roland Lill von der Philipps-Universität ermittelte, welche dreidimensionale Struktur die sechs Proteine des Komplexes aufweisen, der an der Synthese von Eisen-Schwefel-Clustern beteiligt ist. Das Team veröffentlicht seine Ergebnisse in der Wissenschaftszeitschrift „Nature Communications“.
„Enzyme mit Eisen-Schwefel-Clustern sind unersetzlich für zahlreiche essenzielle Lebensprozesse“, erläutert Lill. Beispiele hierfür sind insbesondere die Energiegewinnung in den Mitochondrien, die Vervielfältigung der Erbsubstanz DNA sowie deren Reparatur nach Schädigungen. „Vor kurzem konnte ein Eisen-Schwefel-Protein charakterisiert werden, das bei der zellulären Virenabwehr hilft“, führt der Marburger Hochschullehrer aus. Ist der Prozess der Synthese von Eisen-Schwefel-Clustern gestört, kommt es zu schwerwiegenden neurologischen Erkrankungen.
Die Entstehung von Eisen-Schwefel-Proteinen ist ein mehrstufiger Prozess, der das Zusammenwirken einiger spezialisierter Enzyme erfordert; er findet in den Mitochondrien statt, die als die Kraftwerke der Zelle gelten. Lill und seine Kollegen präsentieren nun drei Kristallstrukturen, „Schnappschüsse unterschiedlicher Stadien des Syntheseprozesses“, wie Lill erläutert. „Aus diesen Daten konnten wir zum ersten Mal herauslesen, wie die verschiedenen Enzyme des Komplexes räumlich zueinander angeordnet sind und wie sie ihre Funktion koordinieren, um aus Eisen und Schwefel einen Eisen-Schwefel-Cluster zusammenzusetzen.“
Dabei blieben auch Überraschungen nicht aus – so fanden die Forscher heraus, dass zwei kleine Proteine, deren Funktion bisher unbekannt war, eher eine steuernde Funktion übernehmen, als dass sie die Clustersynthese direkt unterstützen: Die Proteine ISD11 und ACP stellen vielmehr sicher, dass die Clustersynthese in enger Abstimmung mit dem Energiehaushalt der Zelle geschieht.
Professor Dr. Roland Lill leitet das Institut für Zytobiologie und Zytopathologie der Philipps-Universität und gehört dem Marburger „LOEWE-Instituts für Synthetische Mikrobiologie“ an. Seit dem Jahr 2003 ist der Biochemiker Träger des Leibnizpreises, des am höchsten dotierten deutschen Wissenschaftspreises; einem Ranking der Zeitschrift „Laborjournal“ zufolge zählt er zu den dreißig meistzitierten deutschen Zellbiologen.
Die Publikation ging aus einer engen Kooperation zwischen Lills Arbeitsgruppe und kanadischen Wissenschaftlern von der University of Saskatchewan hervor. Die Deutsche Forschungsgemeinschaft und das hessische Förderprogramm „LOEWE“ unterstützten die Forschungsarbeit finanziell.
Originalveröffentlichung: Michal T. Boniecki, Sven A. Freibert & al.: Structure and functional dynamics of the mitochondrial Fe/S cluster synthesis complex, Nature Communications 2017, DOI: https://doi.org/10.1038/s41467-017-01497-1
Weitere Informationen:
Ansprechpartner: Professor Dr. Roland Lill,
Institut für Zytobiologie und Zytopathologie
Tel.: 06421 28 66449
E-Mail: lill@staff.uni-marburg.de