27.09.2021 Neue Einblicke in Vererbungsprozesse bei Bakterien
Eine multidisziplinäre Studie deckt auf, wie ein molekularer Schalter die Verteilung der bakteriellen DNA steuert
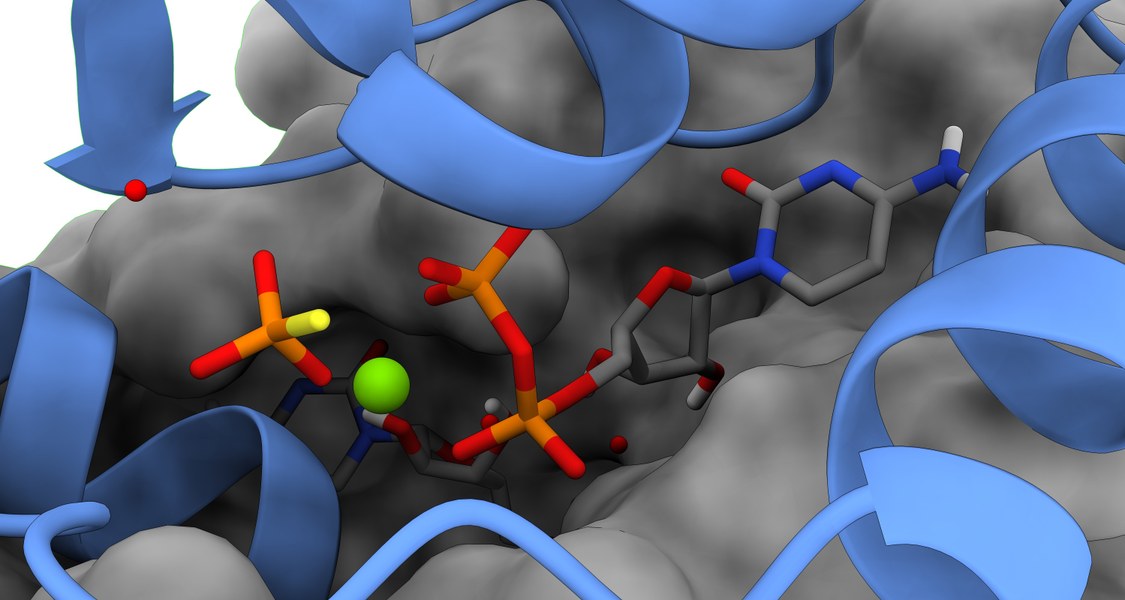
Bei der Teilung einer Zelle müssen beide Tochterzellen einen vollständigen Satz genetischer Informationen erhalten. Im Jahr 2019 entdeckten Forschende um Max-Planck-Fellow Prof. Dr. Martin Thanbichler einen CTP-abhängigen molekularen Schalter, der diesen Prozess steuert. Nun konnte ein internationales Forscherteam zeigen, wie die Hydrolyse von CTP die Dynamik der bakteriellen DNA-Segregation bestimmt. Ihre Entdeckung könnte zur Entwicklung spezifischer Enzyminhibitoren mit potenzieller antimikrobieller Aktivität beitragen.
Nukleotide sind wesentliche Co-Faktoren für eine Vielzahl biochemischer Reaktionen, denn sie sind in der Lage, Energie in Form energiereicher chemischer Bindungen zu speichern. Ihr Abbau (Hydrolyse) treibt die Mehrzahl der energieabhängigen Vorgänge in der Zelle an. Darüber hinaus trägt ihre Hydrolyse dazu bei, Konformationsänderungen in Proteinen auszulösen, die als molekulare Schalter in verschiedenen zellulären Abläufen fungieren. Daher ist die Nukleotid-abhängige Regulierung der Proteinfunktion ein wichtiges Thema in der Biologie.
Eine neue multidisziplinäre Studie unter Leitung von Professor Martin Thanbichler vom Max-Planck-Institut für terrestrische Mikrobiologie und der Philipps-Universität Marburg hat nun eine neue Funktion der Nukleotidhydrolyse bei der bakteriellen DNA-Segregation aufgeklärt.
Bei den meisten Bakterien wird die Verteilung der neu kopierten DNA-Moleküle durch ein dreiteiliges System, das so genannte ParABS-System, gesteuert. Kürzlich hatten die Wissenschaftler entdeckt, dass sein Schlüsselprotein, ParB, eine neue Klasse von nukleotidabhängigen molekularen Schaltern repräsentiert, die das Nukleotid Cytidintriphosphat (CTP) verwenden. Die genaue Rolle der CTP-Hydrolyse blieb jedoch unklar. In ihrer neuen Publikation in der Fachzeitschrift Molecular Cell klären die Autoren nun die nukleotidabhängige Konformationsdynamik von ParB auf und belegen, dass die CTPase-Aktivität von ParB für die Größe und Dynamik der bakteriellen DNA-Teilungskomplexe entscheidend ist.
Im Verlauf der bakteriellen Zellteilung fügen sich Hunderte von ParB-Dimeren zu einem großen DNA-assoziierten Komplex, dem sogenannten Partitionskomplex, zusammen, der als Andockstelle für die DNA-Segregationsmaschinerie dient. Dabei binden sie zunächst an spezifische Sequenzmotive (parS) in den Ziel-DNA-Molekülen. Die CTP-Bindung ermöglicht ihnen dann den Übergang in eine ringförmige Konformation, in welcher sie die DNA lose umfassen und dann von der ursprünglichen Bindungsstelle weggleiten. In der Folge steht die parS-Sequenz wieder für die Aufnahme neuer ParB-Dimere zur Verfügung. Auf diese Weise reicht bereits ein einziges parS-Motiv aus, um Hunderte von ParB-Ringen auf die DNA zu laden.
Die neue Studie zeigt, dass die CTPase-Aktivität von ParB die Größe des Teilungskomplexes bestimmt, indem sie die Verweildauer der ParB-Ringe auf der DNA und damit den Grad ihrer Entfernung von den parS-Motiven limitiert. Die CTP-Hydrolyse führt zur Öffnung der Ringe, wodurch ParB die DNA loslassen und anschließend erneut an ein parS-Motiv binden kann.
Anhand von CTPase-defizienten ParB-Varianten konnten die Wissenschaftler zeigen, dass dieser Prozess für die ParABS-vermittelte DNA-Segregation unerlässlich ist. Fehlt die Fähigkeit zur CTP-Hydrolyse, bildet ParB hyperstabile Ringe, die länger mit der DNA assoziiert bleiben und sich übermäßig stark über diese ausbreiten, was zu schweren Störungen der Verteilung der DNA führt.
ParB ist die erste bisher bekannte Klasse von CTP-abhängigen molekularen Schaltern - alle bisher charakterisierten molekularen Schalter verwenden stattdessen die Purinnukleotide ATP und GTP. Interessanterweise ist die katalytische Domäne von ParB in anderen Proteinen konserviert. "Es ist wahrscheinlich, dass CTP-Bindung und -Hydrolyse die Aktivität verschiedener anderer Proteinfamilien in einer Reihe von unterschiedlichen zellulären Prozessen kontrollieren. Unsere Ergebnisse ebnen somit den Weg zu einem umfassenden Verständnis der Funktionsprinzipien von CTP-abhängigen molekularen Schaltern in der Biologie", sagt Dr. Manuel Osorio-Valeriano, der Erstautor der Veröffentlichung.
Darüber hinaus bietet diese Studie die ersten detaillierten Einblicke in den katalytischen Mechanismus der CTP-Hydrolyse durch ParB-Proteine. Martin Thanbichler ergänzt: "Unsere Ergebnisse könnten zur Entwicklung spezieller Enzymhemmer mit potenzieller antimikrobieller Wirkung führen. Darüber hinaus ermöglichen die neuen Erkenntnisse ein besseres mechanistisches Verständnis der bakteriellen DNA-Segregation und eröffnen so neue Perspektiven für das Design von selbstreplizierenden, minimalen synthetischen Zellen.“ (Pressetext: Virginia Geisel, MPI)