28.05.2025 Vom Müll zum Booster: Wie RNA das Immunsystem antreibt
Forschende entdecken bislang unbekannte Netzwerke aus RNA und Proteinen, die Immunzellen steuern – ein Durchbruch für die RNA-Forschung
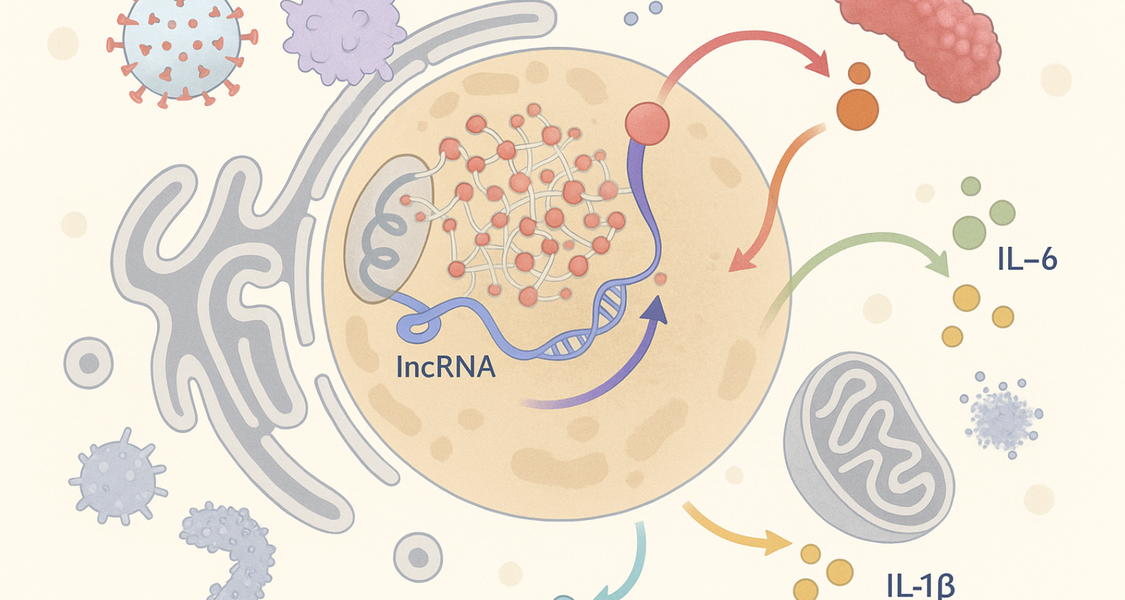
Eine bisher kaum beachtete Klasse von RNA-Molekülen übernimmt offenbar eine Schlüsselrolle in der Immunabwehr gegen bakterielle Infektionen. Forschende um Professor Dr. Leon Schulte vom Institut für Lungenforschung der Philipps-Universität Marburg haben im Fachmagazin Nature Communications einen Atlas veröffentlicht, der zeigt, wie sogenannte lange nichtkodierende RNAs (lncRNAs) gezielt Entzündungsprozesse in Immunzellen beeinflussen. Abschnitte des Erbguts, die lncRNAs codieren, galten lange als „Junk-DNA“ – genetischer Ballast ohne Funktion. Mit einem neu entwickelten Analyseverfahren namens GRADR konnte das Team erstmals umfassend darstellen, wie lncRNAs mit Proteinen in Makrophagen – den „Fresszellen“ des Immunsystems – zusammenwirken. „Wir konnten zeigen, dass viele dieser RNAs direkt in Immunprozesse eingreifen und dabei helfen, Entzündungsreaktionen gezielt zu steuern“, erklärt Schulte. Besonders auffällig: Das RNA-Molekül ROCKI blockiert gezielt einen zellulären „Aus-Schalter“ und fördert so die Entzündungsantwort. „ROCKI und vier weitere, bislang kaum untersuchte lncRNAs beeinflussen zentrale Signalwege der angeborenen Immunantwort“, ergänzt Dr. Nils Schmerer, Erstautor der Studie. Die Forschenden berichten darüber im Fachmagazin Nature Communications (21. Mai 2025, DOI: https://doi.org/10.1038/s41467-025-60084-x)
Neues Werkzeug und offene Daten für die Forschung
Die Ergebnisse basieren auf einer Kombination moderner molekularbiologischer Methoden – darunter RNA-Sequenzierung, CRISPR-Gentechnik und Proteomanalysen. Das daraus entstandene Nachschlagewerk ist öffentlich zugänglich: Über die Webplattform SMyLR können Forschende weltweit gezielt nach Immun-RNAs suchen, ihre Regulation nachvollziehen und passende Proteinpartner identifizieren. „Unsere Ergebnisse zeigen, dass lange nichtkodierende RNAs ein bislang unterschätztes Steuerungselement in der menschlichen Immunabwehr darstellen“, sagt Schulte. Die Erkenntnisse könnten in Zukunft helfen, neue Diagnosemarker oder Therapieansätze für entzündliche Erkrankungen wie Sepsis oder Lungenentzündung zu entwickeln.
Breite Kooperation und Förderung
An der Studie waren zahlreiche Wissenschaftlerinnen und Wissenschaftler des Fachbereichs Medizin der Philipps-Universität Marburg sowie Partnerinstitutionen aus Freiburg, Gießen, Berlin und München beteiligt. Gefördert wurden die Arbeiten von der Deutschen Forschungsgemeinschaft, dem Hessischen Wissenschaftsministerium, der Von Behring-Röntgen-Stiftung und weiteren Unterstützern.
Originalpublikation: Nils Schmerer et al, Nature Communications (21. Mai 2025) https://doi.org/10.1038/s41467-025-60084-x
Zur RNA-Datenbank: http://rna-lab.org/smylr
Kontakt
Prof. Dr. Leon Schulte
Tel.: 06421 28-66852
Mail: leon.schulte@staff.uni-marburg.de
Fachbereich Medizin
Philipps-Universität Marburg