Hauptinhalt
Methoden
Unser Institut nutzt eine Vielzahl modernster molekularer Verfahren zur umfassenden Analyse von Tumoren. Mit Next-Generation-Sequenzierung (NGS) analysieren wir über 400 relevante Gene und erfassen genetische Veränderungen wie Punktmutationen, Genfusionen und Kopienzahlveränderungen. Ergänzt wird dies durch Whole-Exome-Sequenzierung, die das komplette proteincodierende Genom abdeckt, sowie durch Liquid-Biopsy-Panel-Analysen, die zirkulierende Tumor-DNA im Blut minimalinvasiv nachweisen und die Tumordynamik in Echtzeit verfolgen.
Zur räumlichen Untersuchung von Gen- und Proteinexpression nutzen wir fortschrittlichste Technologien wie 10x Visium, CellDIVE und GeoMX. Diese Methoden ermöglichen Einblicke in die Tumormikroumgebung, Immunzellpopulationen und die intratumorale Heterogenität.
Spezialisierte Tests wie EndoPredict für Brustkrebs, HRD-Analysen zur Bewertung von DNA-Reparaturdefekten und der schnelle Idylla-PCR-Test unterstützen die individuelle Therapieentscheidung. Die computergestützte digitale Bildanalyse ergänzt die molekularen Daten durch quantitative Auswertung histologischer Schnitte.
Next-Generation Sequenzierung von Gen-Panels
Unsere Next-Generation-Sequenzierungspanels ermöglichen eine umfassende molekulargenetische Analyse von über 400 krebsassoziierten Genen. Dabei werden punktuelle Mutationen (SNVs), Insertionen und Deletionen (Indels), Kopienzahlveränderungen (CNAs), Genfusionen sowie Exon-Skipping-Ereignisse präzise detektiert. Zusätzlich werden komplexe genomische Biomarker wie Mikrosatelliteninstabilität (MSI) und Tumormutationslast (TMB) bestimmt. Die Bewertung der detektierten Varianten erfolgt mithilfe etablierter Datenbanken zur Ableitung therapeutischer Relevanz. Die Ergebnisse fließen in personalisierte Therapieentscheidungen im interdisziplinären Molekularen Tumorboard ein.
Liquid Biopsy NGS Panel
Das Liquid-Biopsy-Verfahren erlaubt eine minimalinvasive molekulare Charakterisierung von Tumoren durch den Nachweis zirkulierender Tumor-DNA (ctDNA) im Blutplasma. Diese Methode ermöglicht die Echtzeitüberwachung der Tumordynamik, das frühzeitige Erkennen therapieassoziierter Resistenzmechanismen sowie die Langzeitverfolgung molekularer Veränderungen. Die gewonnenen Daten werden bioinformatisch analysiert und unterstützen die individuelle Anpassung der Therapie in laufenden klinischen Studien und Routinediagnostik.
Whole Exome Sequenzierung
Die Whole-Exome-Sequenzierung ermöglicht eine umfassende Analyse aller proteinkodierenden Gene innerhalb des Genoms. Neben der Identifikation von SNVs, Indels und CNAs werden auch komplexe molekulare Marker wie MSI, TMB und HRD detektiert. Die Annotation und Interpretation der genetischen Varianten erfolgt unter Einbeziehung kuratierter Datenbanken, um deren klinische Relevanz zu bewerten. Die WES dient als Grundlage für präzise, personalisierte Therapieentscheidungen und wird im Rahmen des molekularen Tumorboards eingesetzt.
Gene Expression Profiling (whole transcriptome)
Mittels RNA-Sequenzierung wird die Genexpression solider Tumoren umfassend quantifiziert. Die gewonnenen Transkriptomdaten erlauben eine funktionelle Charakterisierung der Tumoren, eine molekulare Subtypisierung sowie die Entwicklung und Validierung prognostischer und prädiktiver Biomarker. Die Ergebnisse werden für Gene-Set-Enrichment-Analysen, multivariate Omics-Integrationen und zur Therapiestratifizierung in klinischen Studien verwendet.
HPV - Humanes Papillomavirus
Die Untersuchung auf humane Papillomviren (HPV) dient der Früherkennung von Infektionen mit Hochrisiko-HPV-Typen, die mit der Entstehung verschiedener Tumoren – insbesondere des Zervixkarzinoms – assoziiert sind. Mithilfe eines einfachen Abstrichs kann eine Infektion frühzeitig detektiert und durch regelmäßige Kontrollen einer Tumorprogression entgegengewirkt werden.
HRD - Homologe Rekombinationsdefizienz
Die Analyse der homologen Rekombinationsdefizienz (HRD) identifiziert Tumoren mit eingeschränkter Fähigkeit zur DNA-Doppelstrangreparatur, wie sie etwa beim Ovarial- oder Mammakarzinom vorkommt. Ein positiver HRD-Status kann eine Sensitivität gegenüber PARP-Inhibitoren anzeigen und dient somit als entscheidender prädiktiver Marker für die Therapieplanung.
Endopredict
EndoPredict ist ein molekulargenetischer Test zur Risikostratifizierung bei Patientinnen mit hormonrezeptorpositivem, HER2-negativem Mammakarzinom im Frühstadium. Der Test basiert auf der Analyse der Expression eines definierten Genpanels in Kombination mit klinisch-pathologischen Parametern. Ziel ist eine präzise Abschätzung des Metastasierungsrisikos innerhalb von zehn Jahren, um eine fundierte Entscheidung über die Notwendigkeit einer adjuvanten Chemotherapie zu ermöglichen.
Idylla
Idylla ist ein vollautomatisiertes qPCR-basiertes Diagnostiksystem, das die schnelle und standardisierte Analyse therapierelevanter Mutationen und Biomarker direkt aus FFPE-Gewebeproben erlaubt. Durch das geschlossene System werden Ergebnisse in kürzester Zeit generiert und ermöglichen so eine unmittelbare Entscheidungsfindung im klinischen Alltag.
10x Visum
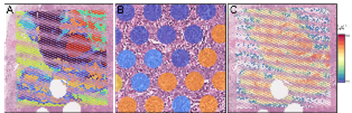
Die 10x Visium Spatial Gene Expression Analysis ermöglicht die präzise Zuordnung von Genexpressionsmustern zur histologischen Struktur von Gewebeproben. Durch die Kombination von Gewebeschnittbildgebung und RNA-Sequenzierung liefert dieses Verfahren eine integrative Perspektive auf die räumliche Organisation der Genaktivität innerhalb des Tumors. So lassen sich differenzielle Expressionsmuster im Kontext von Tumorheterogenität und Mikroumgebung identifizieren. Die Methode eröffnet neue Einblicke in biologische Prozesse der Tumorentwicklung und liefert eine wichtige Grundlage für die Entdeckung räumlich definierter Biomarker.
Genomisches Profiling mit großen Multigen- und Liquid-Biopsy-Panels
Durch den Einsatz großer DNA- und RNA-basierter Multigen-Panels bietet das Institut ein umfassendes molekulares Profiling solider Tumoren. Analysiert werden hunderte Gene, die zentrale onkogene Signalwege abbilden – inklusive Mutationen, Genfusionen, Amplifikationen und komplexer Biomarker wie TMB oder MSI. Ergänzend ermöglicht ein neu etabliertes Liquid-Biopsy-Verfahren die Analyse zirkulierender Tumor-DNA aus Plasmaproben, wodurch molekulare Veränderungen in Echtzeit überwacht und Resistenzmechanismen frühzeitig erkannt werden können.
Die Interpretation der genomischen Daten erfolgt mithilfe der klinischen Softwarelösung MH Guide (Molecular Health), die zugelassene Therapien, potenziell wirksame Substanzen und laufende klinische Studien identifiziert. Diese Plattform unterstützt präzisionsonkologische Entscheidungen im interdisziplinären Tumorboard und liefert einen wichtigen Beitrag zu translationaler Forschung, etwa im Rahmen des MOMENTUM-Registers.Cell Dive - Multiplex-Imaging-Technologie
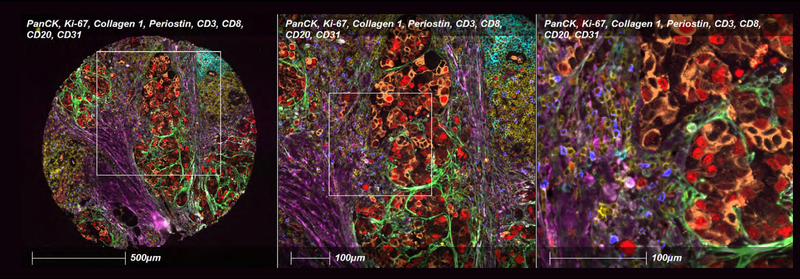
CellDIVE ist eine bildbasierte Multiplex-Immunfluoreszenztechnologie, die die simultane Analyse von bis zu 60 Proteinmarkern in einem einzelnen Gewebeschnitt ermöglicht. Dadurch lassen sich die Zusammensetzung und der Aktivierungsstatus von Immunzellen sowie deren Interaktion mit Tumorzellen präzise kartieren. Die Methode wird unter anderem im Rahmen translationaler Forschungsprojekte zur Charakterisierung der Tumormikroumgebung eingesetzt.
TMA - Tissue Microarrays
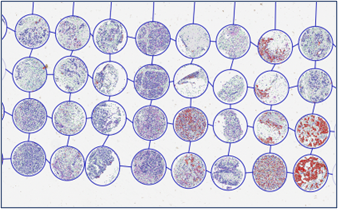
Tissue Microarrays (TMA) ermöglichen die parallele Analyse zahlreicher Gewebeproben unter standardisierten Bedingungen. Kleine Gewebekerne verschiedener Tumoren werden in einem Block vereint und erlauben so hocheffiziente Untersuchungen zur Expression von Biomarkern in großen Kohorten. Die Methode wird insbesondere in der präklinischen Validierung prädiktiver Marker genutzt.
GeoMX - Digital Spatial Profiling
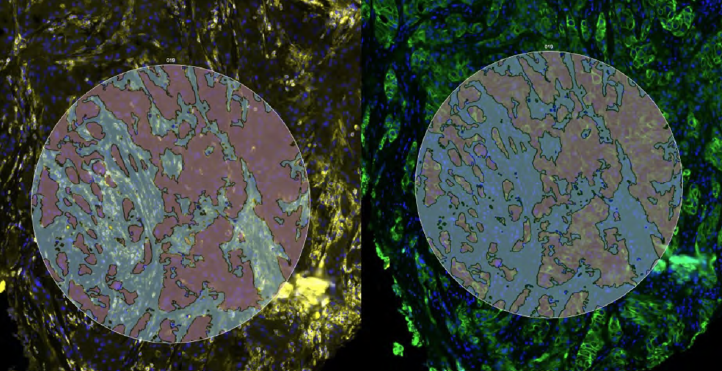
GeoMX ist eine Technologie zur hochauflösenden, multiplexen Analyse der Protein- und RNA-Expression direkt im Gewebekontext. Sie liefert detaillierte Informationen über die räumliche Verteilung molekularer Signaturen innerhalb von Tumorgewebe und dessen Mikroumgebung. Die Methode trägt dazu bei, neue therapeutisch relevante Zielstrukturen und Biomarker im Kontext der Zellinteraktionen zu identifizieren.
Digitale Bildanalyse
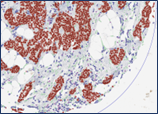
Die computergestützte Bildanalyse erlaubt die quantitative Auswertung histologischer Präparate sowie die präzise Kartierung zellulärer Verteilungen im Tumor und dessen Mikroumgebung. In Kombination mit Multiplextechnologien wie CellDIVE oder EDRMX ermöglicht sie die automatisierte Erkennung funktioneller Muster und unterstützt die Ableitung neuer diagnostischer und prognostischer Marker.