Hauptinhalt
Expression Quantitative Trait Locus- (eQTL-) Analysen
Moderne Technologien haben die integrative Betrachtung des Genoms (DNA) und des Transkriptoms (mRNA) ermöglicht. Hierdurch sind expression quantitative trait locus- (eQTL-) Analysen möglich geworden, bei denen die Allele bzw. Genotypen von genetischen Varianten mit der (quantitativen) Expressionsstärke von Transkripten korreliert werden. Auf diese Weise konnten erstmals Genvarianten identifiziert werden, die die Expressionsstärke von Genen regulieren (s. Abbildung 1).
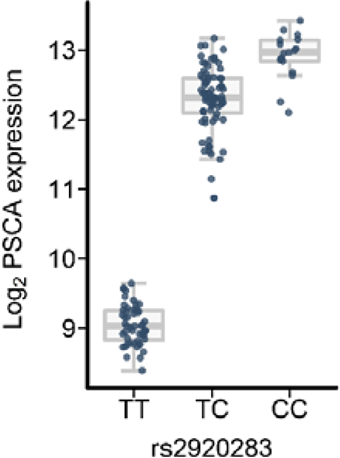
Abbildung 1: Beispielhaft dargestellt ist die Expressionsstärke des Gens PSCA in Abhängigkeit zur Genotypverteilung der Variante rs2223286. Auf der Y-Achse ist die Transkriptmenge von PSCA in der Magenschleimhaut dargestellt. Jeder Punkt entspricht dabei einem Probanden. Diese sind anhand ihrer Genotypausprägung von rs2223286 eingeteilt (X-Achse). Dem Box-Plot ist zu entnehmen, dass Allel C zu einer Expressionsinduktion von PSCA in der Magenschleimhaut führt, wohingegen Allel T zu einer PSCA Expressionsreduktion führt (Heinrichs et al. (2018) Cancer Med). Zuvor konnte die Variante bzw. Allel C in einer genomweiten Assoziationsstudie (GWAS) als Risikofaktor für das Magenkarzinom identifiziert werden (Abnet et al. (2010) Nat Genet). Mit der abgebildeten eQTL-Analyse konnte die GWAS-Risikovariante funktionell charakterisiert werden.
Seit einigen Jahren sind eQTL-Analysen auch genom- und transkriptomweit möglich, wodurch Genregulationsprozesse systematisch erfassbar wurden. Unter maximaler genomischer Abdeckung werden dabei genetische Varianten untersucht und deren Genotypverteilung mit der Expressionshöhe aller erfassbaren Transkripte korreliert.
Ein sehr innovativer eQTL-Ansatz besteht darin, genregulatorische Prozesse nach exogener Stimulation von Zellen zu erfassen (Kim-Hellmuth et al. (2017) Nat Commun, Kim et al. (2014) Nat Commun). Dieses Vorgehen berücksichtigt, dass sich der Einfluss der genetischen Variabilität auf die Genexpression vielfach erst im Rahmen aktivierter zellbiologischer Prozesse nach exogener Stimulation bzw. unter Einwirken von Umweltfaktoren zeigt (s. Abbildung 2).
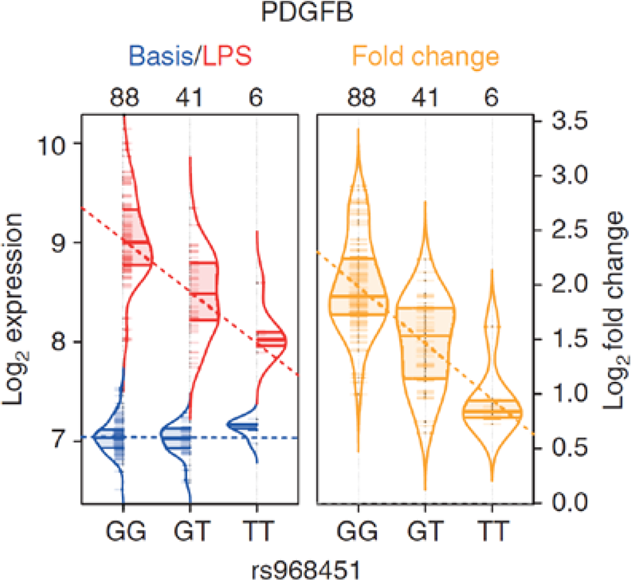
Abbildung 2: Als Beispiel ist ein eQTL in Monozyten dargestellt, der sich ausschließlich nach Stimulation mit Lipopolysacchariden (LPS) zeigt. LPS ist ein Bestandteil der bakteriellen Außenmembran und löst eine Immunantwort in Monozyten über Bindung an Toll-like-4-Rezeptoren (TLR4) aus. Auf der Y-Achse ist die Transkriptmenge des Gens PDGFB in Abhängigkeit der Genotypausprägung der Variante rs968451 dargestellt (X-Achse). In blau zeigt sich kein eQTL-Effekt in unstimulierten Monozyten. Demgegenüber kommt es zu einer PDGFB-Expressionsinduktion in LPS-stimulierten Monozyten (rot). Hierbei zeigt sich ein eQTL-Effekt mit einer stärkeren Expressionsinduktion bei Trägern von Allel G. Im rechten Violin-Plot (gelb) sind die PDGFB-Expressionsdifferenzen vor und nach LPS-Stimulation in Abhängigkeit der Genotypverteilung abgebildet (entnommen aus Kim et al. (2014) Nat Commun).
Solchen Stimulus-spezifischen bzw. exposure eQTLs (e2QTLs) kommt große wissenschaftliche Bedeutung zu. Sie liegen ganz am Beginn eines aktivierten zellbiologischen Prozesses und haben damit großen Einfluss auf die interindividuell unterschiedliche Reaktion des Organismus auf einen Stimulus. Entsprechend konnte eine eigene eQTL-Analyse an unstimulierten sowie mit bakteriellen und viralen Bestandteilen stimulierten Monozyten zeigen, dass 3-18% aller eQTLs Stimulus-spezifisch waren (Kim-Hellmuth et al. (2017) Nat Commun).
Grundsätzlich erlauben eQTL-Analysen aber nicht nur das Erfassen von regulierten Genen und den dazugehörigen Varianten. Sie ermöglichen auch die Identifikation der downstream zum Stimulus wirkenden zellbiologischen Netzwerke bzw. Signalwege, mit denen das Gewebe auf exogene Einflüsse reagiert (s. Abbildung 3).
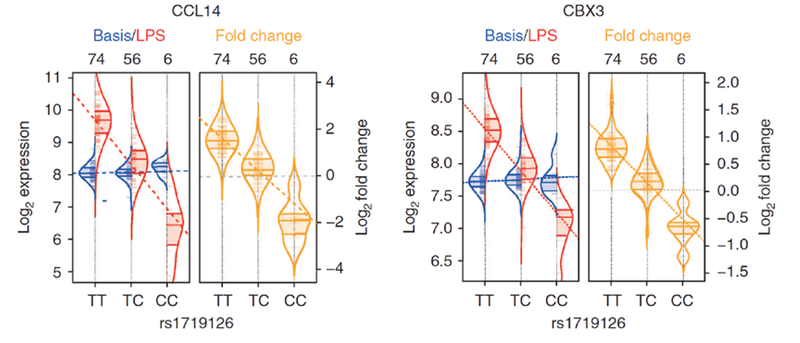
Abbildung 3: Beispielhaft dargestellt sind zwei eQTLs, die ausschließlich in LPS-stimulierten Monozyten aktiv sind (zur Erklärung s. auch Abbildung 2). Der rechte Violin-Plot zeigt einen cis-eQTL zwischen SNP rs1719126 und dem Gen CCL14, die in enger räumlicher Nähe auf Chromosom 17 liegen. Der linke Violin-Plot zeigt einen trans-eQTL zwischen demselben SNP und dem Gen CBX3, das auf Chromosom 7 lokalisiert ist. CCL14 kodiert für ein Chemokin und zeigt im Rahmen der LPS-induzierten Immunantwort starke allelspezifische Expressionsunterschiede. Den nahezu gleichen eQTL-Effekt zeigt die Expression von CBX3, einem Transkriptionsfaktor im Rahmen der Immunantwort (Smallwood et al. (2012) Genome Research). Die eQTL-Analyse legt nahe, dass rs1719126 über CCL14 als “Mediatortranskript“ die Expression von CBX3 reguliert; ein Zusammenhang, der bislang völlig unbekannt war (entnommen Kim et al. (2014) Nat Commun).
eQTL-Analysen kommt darüber hinaus große Bedeutung bei der funktionellen Aufarbeitung von Risikovarianten für multifaktorielle Krankheiten zu, die durch genomweite Assoziationsstudien (GWAS) identifiziert wurden. Zum Beispiel konnte rs968451 in Abbildung 2 als Risikovariante bei einer GWAS zur Primär biliären Zirrhose (PBC) identifiziert werden (Mells et al. (2011) Nat Genet). Aufgrund seiner Lokalisation im intergenen Bereich war der Forschergruppe aber die Benennung des krankheitsrelevanten Gens und des zugrundeliegenden Pathomechanismus nicht möglich. Auch mit den damals vorliegenden eQTL-Daten an nicht-aktivierten Zellen konnte kein PBC-Krankheitsgen in dieser Region favorisiert werden, auf das rs968451 Einfluss ausübt. Die eQTL-Analyse an LPS-stimulierten Monozyten (Kim et al. (2014) Nat Commun) legt nun nahe, dass PDGFB das PBC-relevante Krankheitsgen ist und seine verminderte Expression (Allel T ist das Risikoallel in der GWAS) unter einer TLR4-Aktivierung den Pathomechanismus darstellt.
Das dargestellte eQTL-Konzept ermöglicht zusammenfassend die Identifikation von regulatorischen Varianten und dazugehörigen Genen sowie Signalwegen, die ausschließlich nach Induktion eines zellbiologischen Prozesses durch einen exogenen Stimulus aktiv sind. Ihnen kommt große medizinische Bedeutung zu, da sich die Pathophysiologie vieler Krankheiten erst in Wechselwirkung mit exogenen Einflussfaktoren abbildet. Dabei liegen stimulationsspezifische eQTLs am Beginn des pathophysiologisch relevanten Prozesses und bestimmen die interindividuell unterschiedliche phänotypische Ausprägung.
In Kooperationen mit anderen Forschergruppen beschäftigt sich unsere Arbeitsgruppe gegenwärtig mit e2QTL-Analysen zu metabolischen, inflammatorischen, kardiovaskulären und onkologischen Erkrankungen sowie zu Infektionskrankheiten.
Ansprechpartner
Dr. Benedikt Bürfent
Ausgewählte Publikationen
Kim-Hellmuth S, Bechheim M, Pütz B, Mohammadi P, Nédélec Y, Giangreco N, Becker J, Kaiser V, Fricker N, Beier E, Boor P, Castel SE, Nöthen MM, Barreiro LB, Pickrell JK, Müller-Myhsok B, Lappalainen T, Schumacher J, Hornung V. Genetic regulatory effects modified by immune activation contribute to autoimmune disease associations. Nat Commun 2017 8: 266.
Kim S, Becker J, Bechheim M, Kaiser V, Noursadeghi M, Fricker N, Beier E, Klaschik S, Boor P, Hess T, Hofmann A, Holdenrieder S, Wendland JR, Fröhlich H, Hartmann G, Nöthen MM, Müller-Myhsok B, Pütz B, Hornung V, Schumacher J. Characterizing the genetic basis of innate immune response in TLR4-activated human monocytes. Nat Commun 2014 5: 5236.
Becker J, Wendland JR, Haenisch B, Nöthen MM, Schumacher J. A systematic eQTL study of cis-trans epistasis in 210 HapMap individuals. Eur J Hum Genet 2012 20: 97-101.