Hauptinhalt
AG Becker
Die AG Becker untersucht die Replikation, Morphogenese und Pathogenese hochpathogener Viren wie dem Ebola-Virus (EBOV) und des Marburg-Virus (MARV) sowie von neu auftretenden Coronaviren (CoV) wie dem Sever Acute Respiratory Syndrome Coronavirus (SARS-CoV), dem Middle East Respiratorisches Syndrom Coronavirus (MERS-CoV) und dem pandemischen SARS-CoV2. Die Arbeit der Arbeitsgruppe zielt weiter darauf ab, Virus-Wirt-Interaktionen zu erforschen, da diese Erkenntnisse die Grundlage für die Entwicklung spezifischer antiviraler Therapien bilden.
Neben Experimenten unter BSL4-Bedingungen mit Wildtyp- und rekombinant hergestellten Filoviren werden Modelsysteme auf Basis infektiöser Partikel (infectious-like particles, iVLPs), die eine Virusinfektion unter BSL1-2-Bedingungen nachahmen, verwendet. In der Arbeitsgruppe Becker werden molekulare und biochemische Techniken sowie zellbiologische Methoden wie Immunfluoreszenz, Lebendzellmikroskopie (auch unter BSL4-Bedingungen), konfokale Laser-Scanning-Mikroskopie, hochauflösende Bildgebung wie dSTORM und Elektronenmikroskopie kombiniert angewendet.
Wir sind außerdem an der translationalen Virusforschung beteiligt, beispielsweise an der Charakterisierung von Impfstoffkandidaten in klinischen Phase-1-Studien unter Verwendung von immunologischen Testverfahren. Ziel des Labors ist es außerdem, die antiviralen Mechanismen von Verbindungen zu charakterisieren, von denen bekannt ist, dass sie Virus-Wirt-Interaktionen blockieren.
Forschungsprojekte
Morphogenese und Transport von Filovirus Nukleokapsiden
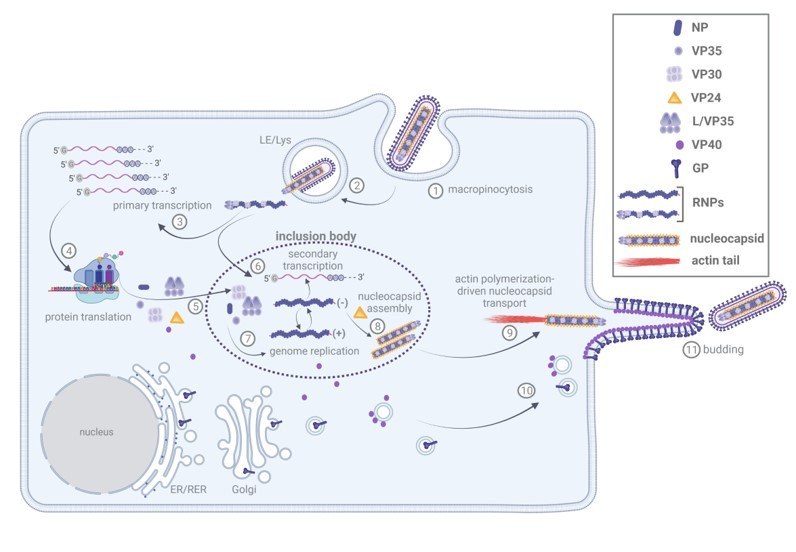
Filoviren sind umhüllt und besitzen ein einzelsträngiges RNA-Genom mit negativer Polarität. Das virale RNA-Genom ist vom Nukleoprotein NP enkapsidiert und assoziiert mit den viralen Proteinen 35 (VP35), VP24 und VP30 sowie mit der viralen Polymerase L, die gemeinsam das Nukleokapsid (NC) bilden.
Die Replikation der Filovirus-Genome findet in Einschlusskörperchen statt, die sich in der Nähe des Zellkerns befinden. Während der Synthese bilden die Genome Ribonukleoprotein (RNP)-Komplexe, die innerhalb der Einschlüsse zu transportkompetenten NCs heranreifen (Dolnik & Becker; PlosPathogens 2022).
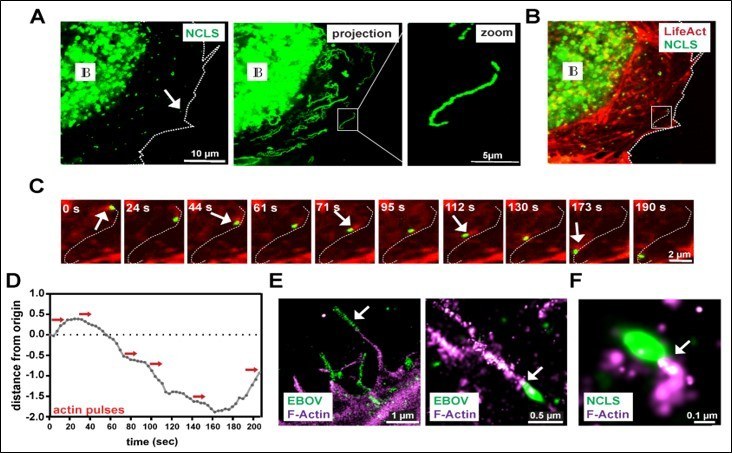
In diesem Projekt untersuchen wir aktuell die räumliche und zeitliche Organisation der filoviralen RNA-Synthese, ihre Verpackung in RNPs und die molekulare Grundlage für die RNP-Reifung zu transportkompetenten NCs. Wir haben festgestellt, dass drei der fünf EBOV-NC-Proteine ausreichen, um transportkompetente nukleokapsidähnliche Strukturen (NCLS) mit derselben Dimension und Struktur wie die authentischen viralen NCs zu bilden. Darüber hinaus charakterisieren wir die zellulären und viralen Faktoren, die an der Rekrutierung der Aktin-polymerisierenden Maschinerie an einem Ende der NCs beteiligt sind, die ihren Transport steuert.
Für weitere Informationen siehe SFB 1021.
Strukturbasiertes Wirkstoffdesign
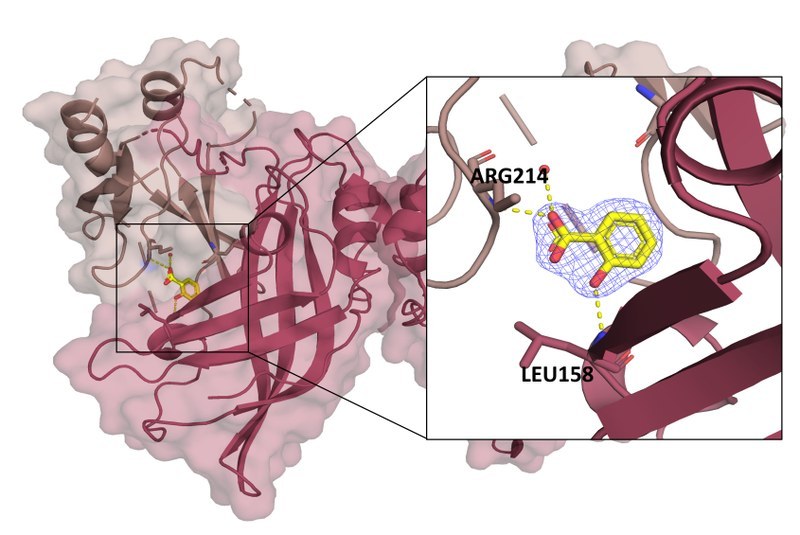
Fragmentbasiertes Wirkstoffdesign (FBDD) oder fragmentbasiertes Lead Design (FBLD) ist ein weit verbreiteter Ansatz zur Identifizierung molekularer Bindemittel, die zu vielversprechenden Hemmstoffen entwickelt werden können. Das Crystal Soaking ist eine hochempfindliche Methode und daher geeignet, schwache Bindemittel aufzuspüren. Ein weiterer Vorteil sind die gewonnenen Strukturinformationen, z. B. wie und wo das Fragment an das Protein bindet. Aufgrund der geringen Anzahl von Atomen und funktionellen Gruppen der Fragmente stellen sie vielversprechende Ausgangspunkte für die Entwicklung von Leitstrukturen dar, so dass nur eine kleine Bibliothek für das Screening benötigt wird. Der wichtigste Vorteil des strukturbasierten Wirkstoffdesigns ist die Entwicklung eines hochspezifischen Hemmstoffs, der das Risiko von Nebenwirkungen bei Patienten senkt.
In diesem Projekt exprimieren wir Ebola-Virus-Proteine in E. coli, reinigen und kristallisieren sie. Die vorgeformten Proteinkristalle werden dann mit kleinen Molekülen getränkt und mit Röntgenstrahlen analysiert. Fragmente, die an das Ebola-Virusprotein im Kristall binden, werden dann für eine weitere Optimierung und Vergrößerung verwendet, um ihre Affinität zum Zielprotein und damit ihre hemmende Wirkung zu verbessern. Neben dem Einweichen von Kristallen werden auch andere biochemische und biophysikalische Methoden eingesetzt, um Bindemittel, Inhibitoren oder PROTACs zu identifizieren. Vielversprechende Kandidaten werden in Zellkulturtests und auch unter BSL-4-Bedingungen mit authentischen Viren getestet.
Wir arbeiten mit zahlreichen anderen Labors aus Marburg, San Diego und Toronto zusammen.
Für weitere Informationen siehe DRUID.
Wirts-gerichtete Virostatika
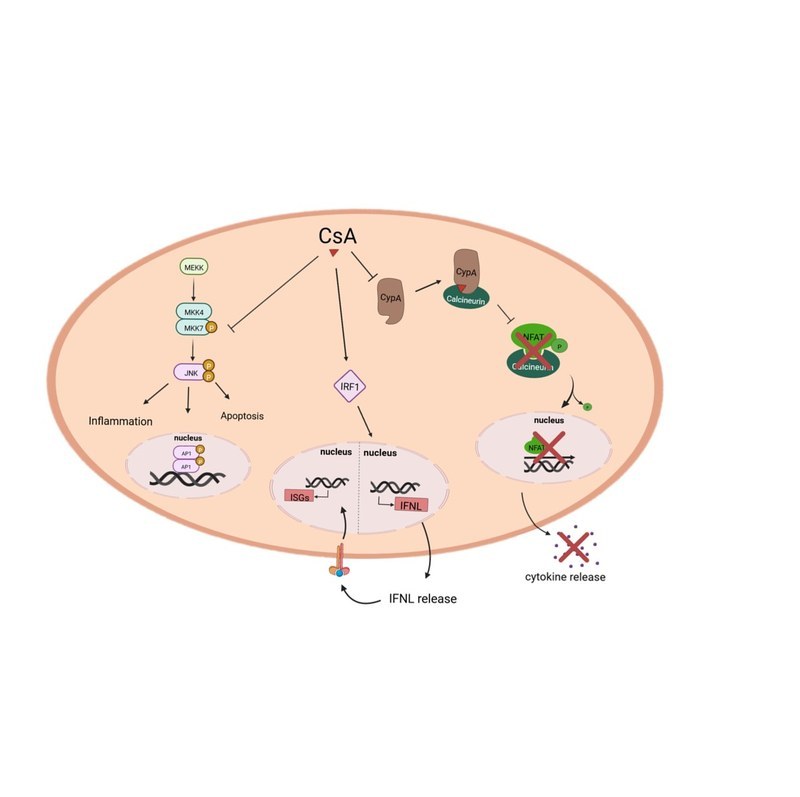
Ein wesentlicher Teil unserer Arbeit in diesem Projekt ist die Erforschung antiviral wirkender Substanzen und ihrem spezifischen Wirkmechanismus gegenüber hochpathogenen Viren. Dabei handelt es sich zum einen um immunsuppressive Substanzen wie Cyclosporin A, die bereits seit Jahrzehnten in der Klinik nach Organtransplantationen verwendet werden und ihre nicht-immunsuppressiven Derivate, welche beide antivirale Eigenschaften gegen verschiedenen Coronaviren zeigten. Wir untersuchen dabei im Detail, wie diese Substanzen die unterschiedlichen Viren hemmen können. Dafür werden unter anderem primäre humane und murine Zellen oder auch verschiedenen ex vivo Zellkultursysteme verwendet. Weiterhin analysieren wir auch die Wirksamkeit von hoch-spezifischen Inhibitoren gegen einzelne virale Proteine und testen diese anschließend in vivo im Hochsicherheitslabor des Instituts für Virologie.
Für weitere Informationen siehe CoroPlan und KFO 309.
Pathogenese und Impfstoffe
Adaptierung von Marburg-Virus an Nagetiere als Modellsystem zur Untersuchung der virale Pathogenese
Marburg-Virus (MARV) verursacht schwere fieberhafte Erkrankungen in Menschen und nicht-menschlichen Primaten. Im Gegensatz dazu entwickeln Nagetiere wie Mäuse oder Meerschweine nur eine vorübergehende nicht-letale Erkrankung. Beim Passagieren von MARV in Meerschweinen kommt es zu adaptiven Mutationen von denen eine im Matrixprotein VP40 und drei in der viralen Polymerase L auftreten. Ziel dieses Projektes ist die Charakterisierung dieser adaptiven Mutationen, um ihren Einfluss auf die Pathogenität zu verstehen.
Zu diesem Zwecke untersuchen wir die genannten Mutationen zunächst mittels Reporterassays in Zellkultursystemen. Im nächsten Schritt erstellen wir unter BSL4 Bedingungen rekombinante Viren, welche einzelne Mutationen oder Kombinationen von Mutationen enthalten. Diese Viren werden im nächsten Schritt sequenziert und mittels Wachstumskurven, Elektronenmikroskopie und weiterer Methoden charakterisiert. Im letzten Schritt erfolgt die Analyse der so erstellten Viren im Tiermodell. Dabei werden Meerschweinchen infiziert und Temperatur, Gewicht und Verhalten der Tiere überwacht und es werden Blutproben im Verlauf genommen. Final erfolgt die Sektion der Tiere und die Viruslast im Blut und in den Organen wird mittels qRT-PCR und TCID50 bestimmt. Außerdem erfolgen eine histologische Untersuchung und Beurteilung der Pathogenität der unterschiedlichen Viren.
Für weitere Informationen siehe auch DZIF.