Hauptinhalt
AG Biedenkopf


Wir sind eine seit Ende 2019 etablierte Nachwuchsgruppe zum Thema Transkriptions- und Replikationsstrategien von hochphatogenen Filoviren am Institut für Virologie (gefördert durch den Forschungscampus Mittelhessen).
Hauptschwerpunkt unserer Arbeiten ist die Regulation der viralen RNA Synthese bei verschiedenen hochpathogenen Viren, wie z.B. Ebola- oder Marburgviren, aber auch MERS-Coronavirus oder SARS-Coronaviren.
Weiterhin etablieren wir für Virusneutralisationstests Pseudovirus/-partikel Surrogatsysteme, die fremde Oberflächenproteine integriert haben sowie Assays zur Austestung von Antiviralen Substanzen gegen hochpathogene Viren unter BSL-4 Bedingungen.
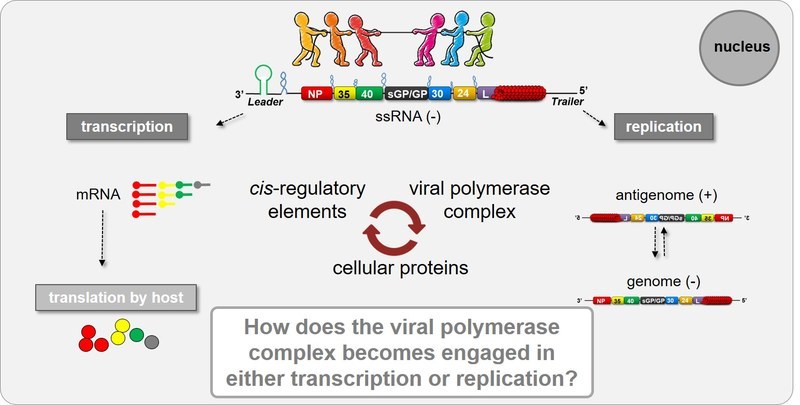
Von großem Interesse für unsere Arbeiten ist, wie, ausgehend vom viralen Genom, zum einen virale mRNAs transkribiert werden (virale Transkription), die von der infizierten Zelle in die viralen Proteinen translatiert werden, oder neue virale Genome in voller Länge (virale Replikation) synthetisiert werden.
Hierbei spielt das Zusammenspiel zwischen cis-aktiven Elementen auf dem RNA-Genom, dem viralen Polymerasekomplex sowie der Interaktion mit zellulären Proteinen eine große Rolle bei der Regulation von viraler Transkription und Replikation regulieren.
Hauptschwerpunkt unserer Arbeiten ist die Rolle der Phosphorylierung von viralen Proteinen bei der Regulation der viraln RNA Synthese durch zelluläre Kinasen und Phosphatasen.
Forschung
Regulation von Transkription und Replikation bei Filoviren
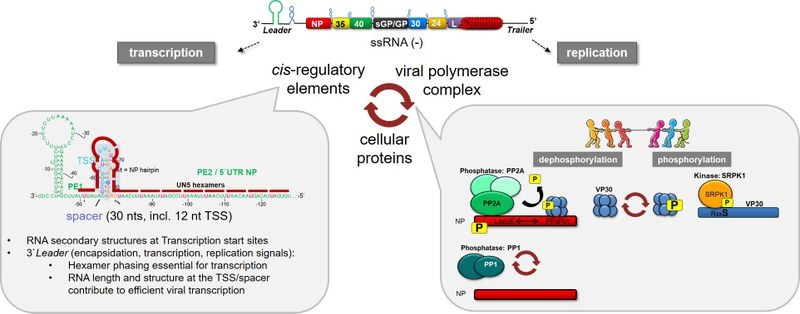
Die Regulation von Transkription und Replikation bei Filoviren wie Marburg- und Ebolavirus ist durch ein Zusammenspiel von cis-aktiven Elementen auf dem RNA-Genom, dem viralen Polymerasekomplex sowie der Interaktion mit zellulären Proteinen reguliert.
Zum einen interessiert uns die Rolle von cis-aktiven Elementen auf dem viralen Genom für die Transkriptionsinitiation. Es handelt sich hier um hochstrukturierte RNA-Elemente des viralen Genoms in den Bereichen der Transkriptionsstartpunkte.
Zum anderen beschäftigen wir uns intensiv mit der Rolle von posttranslationalen Modifikationen (PTMs) von Virusproteinen durch zelluläre Proteine und deren Beitrag zur Regulation der viralen RNA Synthese von Ebola- und Marburgviren.
Besonders interessiert uns hierbei die Phosphorylierung von zwei Nukleokapsidproteinen, dem Nukleoprotein NP und dem virale Transkriptionsfaktor VP30, dessen dynamische Phosphorylierung essentiell zur Regulation der viralen Transkription und Replikation beiträgt.
Die dynamische Phosphorylierung von Ebolavirus VP30 wird durch das Zusammenspiel von zellulären Phosphatasen und Kinasen reguliert, von denen wir einige, wie die Phosphatase PP2A oder die Kinase SRPK1, bereits identifizieren konnten.
Weiterhin interessiert uns die Rolle der Phosphorylierung des Nukleoproteins bei im Verlauf der Virusinfektion (gefördert durch die Von-Behring-Röntgen-Stiftung).
Für weitere Informationen siehe SFB 1021.
Die Rolle der Phosphorylierung von Coronavirus-Nukleokapsidproteinen für den viralen Lebenszyklus
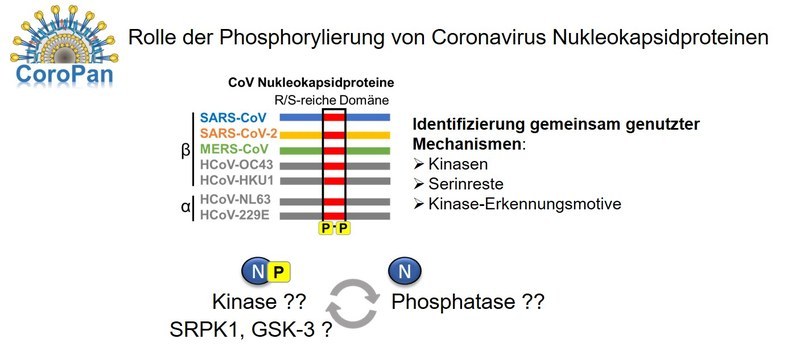
Bei Coronaviren (CoV) spielt das Nukleokapsidprotein N eine Schlüsselrolle bei der RNA Synthese und ist an der Bildung von gRNA als auch bei der diskontinuierlichen Transkription von subgenomischen mRNAs beteiligt.
Die Phosphorylierung von N spielt für dessen Funktionalität eine essentielle Rolle und findet an einem stark konservierten Arginin-Serin-reichen Region statt (R/S Region). Uns interessiert hier besonders,
inwieweit es zwischen unterschiedlichen Coronaviren (α-CoV wie typische Erkältungsviren HCoV 229E oder β-CoV wie SARS-CoV-2 oder MERS-CoV) gemeinsam genutzte konservierte Mechanismen zur Regulation der Virusreplikation durch Phosphorylierung gibt (also z.B. konservierte Phosphorylierungsstellen, konservierte zelluläre Kinasen/Phophatasen).
Entwicklung von Virus-Surrogatsystemen zur Bestimmung von neutralisierenden Antikörpern gegen hochpathogene Viren
Im Fokus dieses Projektes steht die Entwicklung von Virus-Surrogatsystemen als zusätzliche Instrumente zur quantitativen Bestimmung neutralisierender Antikörper oder Seren gegen hochpathogene Viren der Sicherheitsstufe 3 oder 4 (BSL-3/-4). Diese Surrogatsysteme auf Basis von pseudotypisierten oder rekombinanten Viren ermöglichen Untersuchungen zur Neutralisationsaktivität der Proben unter Bedingungen der Biologischen Sicherheitsstufe 2, weil die verwendeten rekombinanten Viren beim Menschen keine Erkrankung auslösen. Dabei werden die Oberflächenproteine von hochpathogenen Viren wie z.B. das Spike-Protein von SARS CoV-2 in replikationsdefiziente Viruspartikel (z.B. Lentiviren) oder in replikationskompetente Viren (rekombinantes Vesikuläres-Stomatitis-Virus [VSV]) eingebaut. Die Pseudoviren werden dann z.B. anstelle des infektiösen SARS-CoV-2 eingesetzt, um Seren von infizierten Patienten oder von geimpften Personen auf ihre Fähigkeit zu untersuchen, eine Infektion zu verhindern.
Nucleosid Booster-Projekt
Dieses Projekt sieht die Testung verschiedener Nukleosidanaloga hinsichtlich ihrer Hemmung der viralen Replikation einer Vielzahl von Viren vor. Es handelt sich hierbei um ein Verbundprojekt mit Partnern der Antiviralen Compound Test Plattform (ACTP) im Rahmen Forschungsbereichs Neu auftretende Infektionskrankheiten des Deutschen Zentrums für Infektionsforschung (DZIF) sowie der Initiative „Drugs for Neglected Diseases Initiative“ (DNDi), die für die Entwicklung der Nukleoside verantwortlich ist. Am Institut für Virologie testen wir die verschiedenen Nukleosidanaloga in einem RT-PCR basierten 96-well System auf ihre Wirksamkeit gegen Filoviren wie Ebolavirus und Marburgvirus, sowie gegen hochpathogene Paramyxoviren wie Nipahvirus. Ziel des Projektes ist es, Nukleosidanaloga zu identifizieren, die eine Breitbandwirkung gegen verschiedene Viren oder Virusfamilien besitzen und diese weiterzuentwickeln.